Nature:双重验证机制确保piRNA通路的精确性
来源:生物探索 2024-10-23 12:29
研究揭示了piRNA通路引导年轻LINE1转座子DNA甲基化的精准控制机制。
转座子(transposons),又称“跳跃基因”,是一类能够在基因组中自行复制并插入到新位置的DNA片段【1】。年轻、活跃的转座子的移动性和复制能力很强,会导致插入基因突变、重排和不稳定性【2】。生殖细胞(如精子和卵子)中需要严格的调控机制来抑制转座子的活动。PIWI-interacting RNA (piRNA) 通路正是其中一种重要机制,它通过引导DNA甲基化来沉默这些转座子,保护基因组完整性【3】。
在小鼠雄性生殖系中,piRNA能够介导MIWI2(PIWIL4)蛋白与年轻转座子转录本结合,启动SPOCD1蛋白介导的DNA甲基化,从而抑制这些转座子的表达和活动【4】。然而,DNA甲基化的精确度必须得到严格控制,避免误作用于正常基因区域而引起基因表达异常。因此,甲基化需要精确定位于转座子序列,但这种精确识别机制背后的分子机制仍不完全清楚。
近日,英国爱丁堡大学Dónal O’Carroll团队在Nature上发表了题为Two-factor authentication underpins the precision of the piRNA pathway的文章,揭示了piRNA通路引导年轻LINE1转座子DNA甲基化的精准控制机制。

piRNA指导的MIWI2通常是在转座子转录已经启动之后与其结合【5】,这表明,在MIWI2结合之前,可能已经存在某种染色质标记或蛋白复合物参与了对年轻转座子的初步识别。作者首先通过ChIP-seq(染色质免疫共沉淀-测序)技术分析了小鼠胚胎发育不同阶段(E13.5到E19.5)中年轻与年老转座子的染色质标记模式,发现年轻的LINE1转座子家族(如L1Md_T, L1Md_Gf, L1Md_A)在E13.5时就被H3K4me3和H3K9me3双重标记,而这一标记在piRNA启动前就已存在。
这种标记的存在提示可能有其他蛋白(如SPIN1)能够识别这些修饰并参与转座子的初步识别。SPIN1能够与特定染色质修饰(如H3K4me3)结合【6】,SPOCD1则是piRNA通路中与MIWI2相互作用并启动DNA甲基化的关键因子【7】。作者假设:SPOCD1与SPIN1之间的相互作用可能将转座子的染色质标记信息传递至DNA甲基化通路,形成连接染色质修饰与基因沉默的分子桥梁。
为了验证这一假设,作者通过免疫共沉淀、质谱分析等实验,发现SPOCD1的328-347氨基酸区域能够与SPIN1的TLD3结构域直接相互作用。接着,他们构建了SPOCD1突变体(Spocd1ΔSPIN1)小鼠,该突变体无法与SPIN1结合。突变体小鼠显示出严重的精子发生障碍,并且年轻LINE1转座子的DNA甲基化水平显著下降,这表明SPOCD1-SPIN1相互作用在piRNA引导的DNA甲基化中具有关键作用。
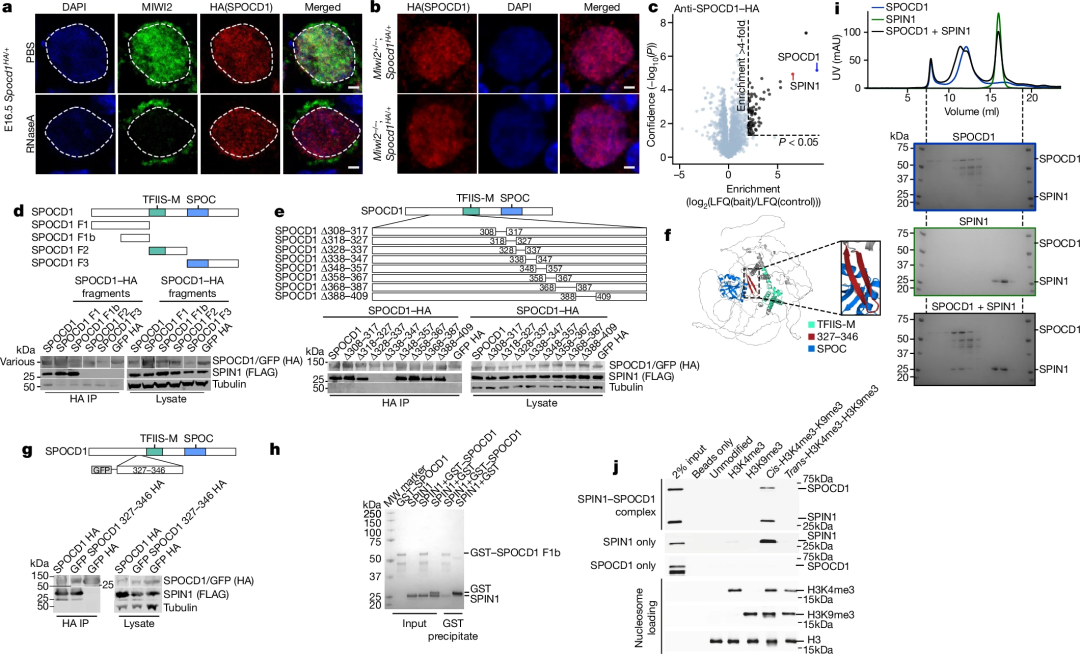
图1:展示了小鼠胚胎期(E16.5)睾丸切片中MIWI2和SPOCD1的定位(Credit: Nature)
最后,研究团队设计了时序依赖性实验,探究SPIN1在piRNA启动前(E13.5)是否已经定位于年轻转座子上。他们利用免疫染色与CUT&Tag(染色质免疫标记与目标分析)技术,验证了SPIN1在MIWI2表达前就已结合在H3K4me3与H3K9me3共修饰的年轻LINE1转座子上,表明SPIN1能够在MIWI2结合之前对转座子进行初步识别。
综上所述,文章揭示了piRNA引导的DNA甲基化过程中对年轻转座子的精确识别机制。研究表明,SPIN1能够通过识别H3K4me3-K9me3双重标记的年轻LINE1转座子,并通过与SPOCD1的相互作用介导初步识别。这一过程是piRNA指导MIWI2识别新生转录本的前提条件,即DNA甲基化启动前的第一步验证机制。而MIWI2与新生转录本的结合则是第二步验证机制,从而形成双重验证机制以确保DNA甲基化的精确性。
两步验证机制提高了对转座子的识别精确性,避免了异位甲基化对基因组功能的影响,还为理解转座子的调控机制提供了新的视角。未来的研究可以进一步探讨在其他类型的转座子(如IAP)中是否存在类似的识别与调控机制,并揭示参与转座子识别的其他蛋白与染色质标记,从而更全面地理解piRNA通路在基因组保护中的作用。
参考文献
1. Kazazian, H. H. (2004). Mobile elements: Drivers of genome evolution. Science, 303(5664), 1626-1632.
2. Aravin, A. A., Sachidanandam, R., Girard, A., Fejes-Toth, K. & Hannon, G. J. Developmentally regulated piRNA clusters implicate MILI in transposon control. Science 16, 744–747 (2007).
3. Ozata, D. M., Gainetdinov, I., Zoch, A., O’Carroll, D. & Zamore, P. D. PIWI-interacting RNAs:small RNAs with big functions. Nat. Rev. Genet. 20, 89–108 (2019).
4. Schöpp, T. et al. TEX15 is an essential executor of MIWI2-directed transposon DNA methylation and silencing. Nat. Commun. 11, 3739 (2020).
5. De Fazio, S. et al. The endonuclease activity of Mili fuels piRNA amplification that silencesLINE1 elements. Nature 480, 259–263 (2011).
6. Du, Y. et al. Structural mechanism of bivalent histone H3K4me3K9me3 recognition by the Spindlin1/C11orf84 complex in rRNA transcription activation. Nat. Commun. 12, 949 (2021).
7. Zoch, A. et al. SPOCD1 is an essential executor of piRNA-directed de novo DNA methylation.Nature 584, 635–639 (2020).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


